메탄가스 전환 미생물촉매 개량을 위한 플라스미드 복제 시작점 예측


Copyright © 2023 by the New & Renewable Energy
This is an Open Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Abstract
Methane is the second most emitted greenhouse gas after carbon dioxide. Despite lower emissions than those of carbon dioxide, methane receives significant attention owing to its more than 20-fold higher global warming potential. Consequently, the importance of research on methanotrophic bacteria, microorganisms capable of converting methane gas into high-value materials, is increasingly emphasized. In the case of methanotrophic bacteria, knowledge on episomal plasmids that can be used for genetic engineering remains lacking, which poses significant challenges to the engineering process. The replication origin sequences of natural plasmids within methanotrophic bacteria have been predicted through in silico methods. The basic characteristics of the replication origin, such as a high A/T ratio, repetitive sequences, and proximity to proteins related to replication, have been used as criteria for identifying the replication origin. As a result, a region with a sequence of 18 base pairs repeated eight times could be identified. The putative replication origin sequence thus identified generally takes the form of iterons, but it also possesses unique features such as the length of the gap between iterons and the repetition of identical iteron sequences. This information can be valuable for future design of episomal plasmids applicable to methanotrophs.
Keywords:
Methane, Methanotroph, Plasmid, Replication origin키워드:
메탄, 메탄자화균, 플라스미드, 복제시작점1. 서 론
메탄가스는 그 배출량에 있어 이산화탄소에 이어 두 번째로 많이 배출되는 온실가스이다.[1] 메탄가스가 가진 global warming potential은 이산화탄소보다 높은 것으로 알려져있다. 메탄가스의 global warming potential은 20년을 기준으로 생각하였을 때는 이산화탄소보다 84배 높고 100년을 기준으로 생각하였을 때는 28배 정도높은 것으로 생각된다.[2,3] 뿐만 아니라 미국해양대기청의 Mauna Loa 관측소의 기록에 따르면 메탄가스는 산업혁명이전 700 ppb에서 현재 1900 ppb 수준으로 빠르게 증가하고 있다. 이러한 사실들로 인해 이산화탄소보다 적은 배출량에도 불구하고 메탄가스에 대한 세계적인 관심은 계속 높아지고 있다. 특히 2021년 유엔 당사국 총회(COP26)에서는 2030년까지 메탄의 배출량을 30%이상 감축하는 것을 목표로하는 글로벌 메탄 서약(Globla Methane Pledge) 프로그램을 추진하기로 결정하였다.[4]
이에 발맞추어 메탄가스를 고부가 물질로 전환하는 기술에 대한 관심 또한 계속 높아지고 있다. 특히 메탄자화균을 이용하여 고부가 물질을 생산하는 기술은 상온 상압에서 반응이 이루어질 수 있다는 점에서 화학전환 공정에 비해 장점을 가지로 있다. 메탄자화균을 이용하여 젖산, 숙신산, 2,3-butanediol, squalene 뿐 만아니라 생분해성고분자인 polyhydroxybutyrate(PHB)에 이르기까지 다양한 물질을 생산하는 것이 가능하며 genetic engineering을 통해 그 생산성을 높이려는 시도가 계속되고 있다.[4~9] 그러나 메탄자화균의 경우 아직까지 미생물 촉매를 개선하기 위한 유전공학적 도구들이 충분히 개발되어 있지 못한 것이 현실이다. 특히 외부 유전자를 전달하기위한 episomal plasmid vector의 경우 종에 따라 그전에 알려진 vector들이 전혀 사용되지 못하는 경우가 많다.[10]
본 연구는 이러한 메탄자화균의 기술적 한계를 극복하기 위한 실마리를 제공하기 위해 진행되었다. Plasmid vector를 설계함에 있어 가장 중요한 정보는 genome의 바깥에서 plasmid가 독립적으로 존재할 수 있도록 해주는 복제시작점 서열이다. Methylomonas sp. DH-1의 경우 277 kb의 길이를 지니는 episomal plasmid를 가지고 있는 것으로 보고되었다.[11] 이 plasmid의 복제시작점의 경우 이전에 보고된 바 없으며 그 서열이 밝혀지는 경우 메탄자화균을 위한 새로운 episomal plasmid를 설계함에 있어 중요한 정보를 제공할 수 있을 것으로 생각된다. 본 연구를 in-silico 분석을 통하여 DH-1 strain 내부에 존재하는 episomal plasmid의 복제시작점을 예측하였다.
2. 재료 및 방법
DNA 서열의 경우 GenBank(https://www.ncbi.nlm.nih.gov/genbank)에 공개된 서열(Methylomonas sp. DH-1: accession number CP014361, Methylomicrobium album BG8: accession number CM001476)을 사용하였다. Plasmid 내 유전자 서열 예측은 Prodigal V2.6.3을 사용하였다. 단백질 서열의 유사성 확인을 위해 BLAST 프로그램을 이용하였으며 data base로 GenBank nr database 및 PFAM database를 활용하였다. DNA 서열의 유사도 확인을 위하여 BLASTN 프로그램을 사용하였다.
3. 분석결과
3.1 Plasmid의 복제시작점(oriV: origin of vegetative replication) 분석 전략
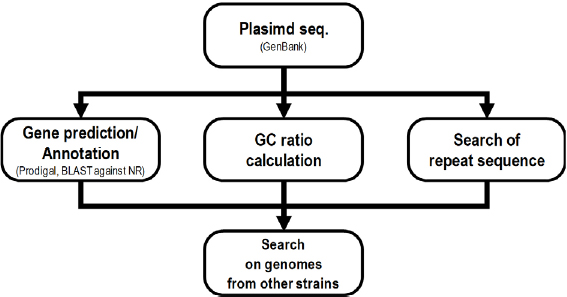
Plasmid의 복제시작점의 경우 replication initiation proteins(RepA, RepB etc.) 및 plasmid stabilization proteins(StbA) 존재하는 위치 가까이에 존재할 가능성이 높은 것으로 알려져 있다.[12] Plasmid 내에 존재하는 단백질 서열을 검색하여 replication initiation protein들과 plasmid stabilization protein의 위치를 먼저 파악하였다. 또한 복제원점은 유전체 및 plasimd 전체에서 AT 비율이 높게 유지되는 것으로 알려져 있기 때문에 plasmid내에 non-coding region의 GC ratio를 계산하였다.[12] oriV의 경우, iteron과 같은 반복 서열이 존재하는 것으로 알려져 있어 repeat sequence 검색을 수행하였다. 위 정보를 종합하여 plasmid의 복제 원점을 예측하고 타 균주의 유전체 정보에서 서열이 유지되는 지 확인하는 방식으로 oriV 서열 예측을 진행하였다(Fig. 1 참조).
3.2 Strategy for the prediction of plasmid 복제시작점 in Methanotrophs
GenBank(https://www.ncbi.nlm.nih.gov/)로부터 Methylomonas sp. DH-1의 plasmid의 완전 해독된 염기서열(accession number CP014361)을 확보하였다. 확보된 염기서열을 기반으로 Prodigal V2.6.3 프로그램을 활용하여 유전자 annotation을 진행하였으며 그 결과 247개의 CDS(protein coding sequence)가 예측되었다. 각 CDS를 GenBank nr database 및 PFAM database를 활용하여 기존 알려진 유전자들과 유사성 검색 수행하여 그 기능을 예측하였다. 그 중 replication/conjugation과 직접적인 연관이 있는 유전자는 17개로 Table 1에 정리하였다.
복제시작점의 경우 AT 비율이 상대적으로 높은 것으로 알려져 있다. CDS 사이 gap에 해당하는 염기서열의 길이가 400 bp 이상인 부분에 대하여 GC ratio를 계산하였다. Plasmid의 전체 GC ratio는 51.66% 였으며, 91,232~92,018 base에서 가장 낮은 GC ratio(37.61%)를 보였다(Table 2 참조). 이외에도 107 kbase, 117 kbase, 207 kbase 근처에서 평균보다 10% 낮은 ratio를 보이는 것을 확인하였다.
각 gap에서 10 mer 이상의 internal repeat sequences를 검색한 결과, gap5, gap6, gap10, gap12, gap17, gap21, gap23에서만 repeat sequences가 발견되었다. 이 중, gap5, gap12, gap17, gap21 부위에서 근거리 repeat sequences가 존재함이 관찰되었다.
또한 gap sequences들을 기 보고된 Methylomonas genus 유전체 서열 중 plasmid replication 관련 유전자를 포함하는 contig들과 비교한 결과, gap6, gap12, gap16에서의 각각 15 bp, 19 bp, 11 bp 염기서열이 높은 유사성(91.9%>)을 보이는 것으로 확인되었다. 특히, gap12는 다른 것들보다 좀 더 긴 부위에서 보존 되어 있음이 확인되었다(Fig. 2 참조).
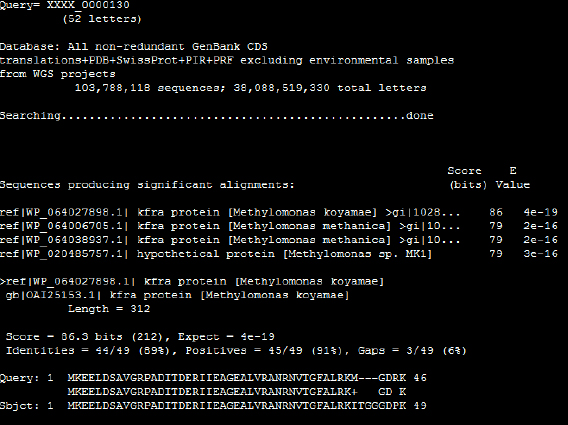
그러나 DH-1의 plasmid 내부에는 plasmid replication에 필요한 repABC 유전자가 암호화되어있지 않았다. 다만, 가장 AT-rich한 영역인 91,232~92,018의 upstream에는 DNA replication에 관여하는 것으로 알려진 kfrA gene(XXXX_0000130)이 존재하는 것이 확인되었다(Table 1 참조). Methylomonas의 kfrA gene은 300여개의 amino acid를 coding하는 것으로 알려져 있지만, XXXX_0000130 유전자는 52 개의 amino acid로 구성되어 있다. 이 52개의 아미노산은 Blast 분석 결과 기존에 보고된 kfrA의 C-terminal을 coding하고 있었다(Fig. 3 참조).
이러한 사실들을 기반으로 생각하였을 때 본래의 kfrA 유전자가 transduction 등의 genetic event로 인하여 분절된 것으로 추정할 수 있다(Fig. 4 참조).

The genetic structure of DNA sequences near a putative oriV loaction. kfrA gene (yellow), hypothetical protein (grey), transposon related genes (red), oriV (green)
온전한 크기의 kfrA gene(XXXX_0000286)은 263,686~264,591에 위치하고 있으나 이 유전자의 downstream에 위치한 gap sequences(gap26)의 GC ratio가 46%로 낮지 않으며, repeat sequence가 존재하지 않는다. 또한, 이 영역의 서열은 타 균주에서 보존되어 있지 않았다. 또한, 91,232~92,018 영역에서 보존된 sequence에는 repeat sequence(AAGT(A)AATAAA)를 포함하고 있는 것이 확인되었다(Fig. 2 참조).
이외에도 본 plasmid에는 conjugation 관련된 유전자들(tra genes)을 다수 포함하고 있는 것이 확인되었다(Table 1 참조). Conjugation을 위하여서는 일반적으로 oriV 이 아닌 oriT를 요구하는 것으로 알려져있다. oriT는 inverted repeat을 포함하고 있어 stem-loop 구조를 이루는 것으로 알려져 있기 때문에 inverted repeat finder를 활용하여 분석을 진행하였다. 그 결과, 두 영역에서 20 mer 수준의 repeat이 발견되었다(Table 3 참조).
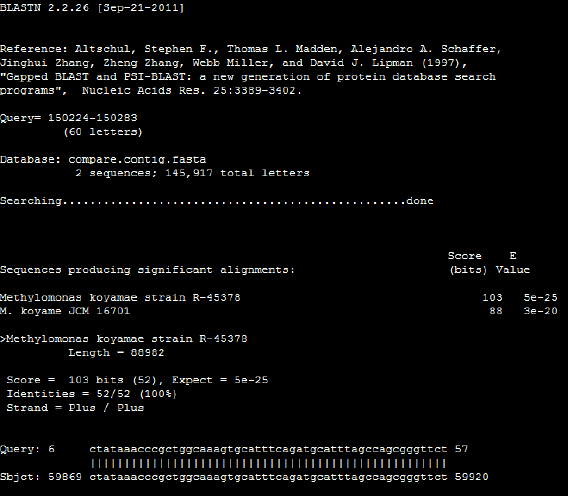
이들 inverted repeats 영역을 타 균주에서 유사성 검색을 수행하였다. 그 결과, 150,234~150,253을 포함한 서열이 다른 두 균주에서 잘 보존되어 있음을 확인할 수 있었다(Fig. 5, 6 참조). 또한, 150,234~150,253의 주위에는 plasmid stabilization protein stbA 유전자가 위치하고 있었다.
본 결과를 종합하여 볼 때, 91,232~92,018의 영역과 kfrA gene(XXXX_0000286)이 plasmid replication에 관여할 것으로 예상되며, 150,234~150,253 영역은 conjugation의 복제 원점으로 추정된다.
3.3 Methylomicrobium album BG8의 플라스미드 분석
기본적으로 DH-1 strain과 동일한 전략을 사용하였다. GenBank로부터 Methylomicrobium album BG8의 plasmid의 완전 해독된 염기서열(accession number CM001476) 확보하여 Prodigal V2.6.3 프로그램을 활용하여 유전자 예측 수행하였다. 그 결과, 49개의 CDS(protein coding sequence)를 확보하였다. 각 CDS를 GenBank nr database 및 PFAM database를 활용하여 기존 알려진 유전자들과 유사성 검색 수행함. 그 결과, 본 plasmid 내에 8개의 replication 관련된 유전자가 존재함을 확인하였다(Table 3 참조).
CDS 사이 gap에 해당하는 염기서열의 길이가 400 bp 이상인 region에 대하여 GC ratio를 계산하였다(Table 4 참조). 본 plasmid 전체의 GC ratio는 52.60% 였으며, Table 4에서 보듯 45,226~45,996 base에서 가장 낮은 GC ratio(33.59%)를 보였다.
각 gap에서의 internal repeat을 조사한 결과, gap2 및 gap5에서 interal repeat이 존재하는 것을 확인하였음. 특히, gap5의 경우, 서열 CCCCTAAACAGTGATAT 이 8번 반복되는 것을 확인할 수 있었음. 이 반복 서열은 Methylomicrobium agile strain ATCC 35068의 유전체 초안 서열에서도 존재(CCCCTAAACAGTGA CAT)하는 것을 확인할 수 있었으며, 역시 근접한 거리에서 5번 반복되는 것을 확인할 수 있었다(Fig. 7 참조).
gap5 주위에 repB, kfrA, repA gene이 위치하고 있어 iteron containing plasmid에서 볼 수 있는 replicon의 모습을 갖추고 있는 것이 확인되었다(Fig. 8 참조)
4. 결과 및 토의
메탄자화균 중 Methylomonas sp. DH-1, Methylomicrobium album BG8 두 strain 내에 존재하는 plasmid의 복제시작점이 예측되었다. 이전에 알려진 복제시작점의 특성에 대한 정보를 기반으로 하여 복제시작점을 가지고 있을 확률이 높은 서열을 찾아내었다. 그 결과 gap5에 해당하는 서열에서 18개의 nucleotide 서열이 8번 반복되는 것이 확인되었다. 이러한 서열의 반복은 direct repeat의 성격을 가지고 있다. 이러한 복제시작점 주변의 반복서열은 RepA 단백질이 결합하는 자리로 생각되며 iteron이라 불린다. 보통 iteron들은 20 bp 안팎의 길이를 가지는 것으로 알려져 있으며 DH-1 내부 plasmid의 iteron 또한 18 bp의 길이를 가지고 있다.[13,14] 이러한 특성들은 그동안 알려진 그동안 theta type replication을 하는 것으로 알려진 다른 plasmid들과 유사하다. 그러나 이번에 찾아진 DH-1 plasmid의 예상 복제시작점 서열의 경우 그동안 알려진 다른 복제시작점과는 몇 가지 다른 특성을 지니고 있는 것으로 생각된다. pPS10의 경우를 제외한 대부분의 알려진 iteron 서열의 경우 origin 안에서 완전히 같은 서열로 반복되지는 않는다. 그러나 이번에 찾아진 DH-1 plasmid의 iteron 서열의 경우 그 nucleotide 서열이 완전히 일치한다. 또한 iteron서열들은 일반적으로 11 bp의 gap을 두고 배치되어 있는 것으로 알려져 있는데 이는 DNA 이중나선 구조의 회전 주기와도 어느 정도 일치한다.[12] 그러나 이번에 찾아진 서열의 경우 7개의 interon 사이의 gap 중 4개의 gap에서 5 bp의 서열[TTCA(C/T)]이 반복되는 것이 관찰되었다. 5 bp가 DNA 회전 주기의 절반정도인 것을 생각하였을 때 그동안 알려진 RepA 결합과 다른 형태를 지닌 결합일 가능성이 있다. 새롭게 찾아진 서열이 iteron 형태인 것을 보았을 때 plasmid 복제 방식은 theta-type이거나 strand displacement방식일 것으로 생각된다. 가장 특이한 부분은 RepA 단백질이 plasmid내에 존재하지 않는 다는 점이다. 모균주의 DH-1 strain의 genome에서도 RepA 단백질과 유사한 단백질은 찾아지지 않았다. Plasmid 내에 존재하는 다른 DNA binding protein이 그 역할을 대신할 가능성도 있으나 이에대해서는 추가적인 연구가 필요할 것으로 생각된다.
본 논문이 제시하는 결과는 향후 메탄자화균에 필요한 episomal plasmid를 설계함에 있어 유용한 정보가 될 수 있을 것으로 생각된다. 다만 모든 분석이 in silico에서 진행되었기 때문에 in vivo에서 이 서열이 제대로 작동함을 증명하는 추가적인 실험이 필요할 것으로 생각된다.
References
- Ritchie, H., Rosado, P., and Roser, M., 2020, “Greenhouse gas emissions”, Our World in Data, https://ourworldindata.org/greenhouse-gas-emissions, .
- Intergovernmental Panel on Climate Change (IPCC), 2007, “Chapter 2: Changes in atmospheric constituents and radiative forcing”, In: Climate Change 2007: The Physical Science Basis. Contribution of Working Group I to the Fourth Assessment Report of the Intergovernmental Panel on Climate Change, Cambridge University Press, UK.
-
Intergovernmental Panel on Climate Change (IPCC), 2014, “Chapter 8: Anthropogenic and natural radiative forcing”, In: Climate Change 2013: The Physical Science Basis: Contribution of Working Group I to the Fifth Assessment Report of the Intergovernmental Panel on Climate Change, Cambridge Uninersity Press, UK.
[https://doi.org/10.1017/CBO9781107415324]
- Global Methane Pledge, Accessed 7 January 2023, https://www.globalmethanepledge.org/, .
-
Henard, C.A., Smith, H., Dowe, N., Kalyuzhnaya, M.G., Pienkos, P.T., and Guarnieri, M.T., 2016, “Bioconversion of methane to lactate by an obligate methanotrophic bacterium”, Scientific Reports, 6, 21585.
[https://doi.org/10.1038/srep21585]
-
Lee, J.K., Kim, S., Kim, W., Kim, S., Cha, S., Moon, H., Hur, D.H., Kim, S.Y., Na, J.G., and Lee, J.W., et al., 2019, “Efficient production of D-lactate from methane in a lactate-tolerant strain of Methylomonas sp. DH-1 generated by adaptive laboratory evolution”, Biotechnology for Biofuels, 12, 234.
[https://doi.org/10.1186/s13068-019-1574-9]
-
Nguyen, D.T.N., Lee, O.K., Hadiyati, S., Affifah, A.N., Kim, M.S., and Lee, E.Y., 2019, “Metabolic engineering of the type I methanotroph Methylomonas sp. DH-1 for production of succinate from methane”, Metabolic Engineering, 54, 170-179.
[https://doi.org/10.1016/j.ymben.2019.03.013]
-
Kang, C.K., Jeong, S.W., Jo, J.H., Park, J.H., Kim, M.S., Yang, J.E., and Choi, Y.J., 2021, “High-Level squalene production from methane using a metabolically engineered Methylomonas sp. DH-1 strain”, ACS Sustainable Chem. Eng., 9(48), 16485-16493.
[https://doi.org/10.1021/acssuschemeng.1c06776]
-
Helm, J., Wendlandt, K.D., Jechorek, M., and Stottmeister, U., 2008, “Potassium deficiency results in accumulation of ultra-high molecular weight poly-β-hydroxybutyrate in a methane-utilizing mixed culture”, J. Appl. Microbiol., 105(4), 1054-1061.
[https://doi.org/10.1111/j.1365-2672.2008.03831.x]
-
Nguyen, A.D., Hwang, I.Y., Lee, O.K., Kim, D., Kalyuzhnaya, M.G., Mariyana, R., Hadiyati, S., Kim, M.S., and Lee, E.Y., 2018, “Systematic metabolic engineering of Methylomicrobium alcaliphilum 20Z for 2,3-butanediol production from methane”, Metabolic Engineering, 47, 323-333.
[https://doi.org/10.1016/j.ymben.2018.04.010]
-
Nguyen, A.D., Hwang, I.Y., Lee, O.K., Hur, D.H., Jeon, Y.C., Hadiyati, S., Kim, M.S., Yoon, S.H., Jeong, H., and Lee, E.Y., 2018, “Functional analysis of Methylomonas sp. DH-1 genome as a promising biocatalyst for bioconversion of methane to valuable chemicals”, Catalysts, 8(3), 117.
[https://doi.org/10.3390/catal8030117]
-
Novick, R.P., 1987, “Plasmid incompatibility”, Microbiol Rev., 51(4), 381-395.
[https://doi.org/10.1128/mr.51.4.381-395.1987]
-
Solar, G.D., Giraldo, R., Ruiz-Echevarría M.J., Espinosa, M., and Díaz-Orejas, R., 1998, “Replication and control of circular bacterial plasmids”, MMBR, 62(2), 434-464.
[https://doi.org/10.1128/MMBR.62.2.434-464.1998]
-
Chattoraj, D.K., 2000, “Control of plasmid DNA replication by iterons: no longer paradoxical”, Mol Microbiol., 37(3), 467-476.
[https://doi.org/10.1046/j.1365-2958.2000.01986.x]